Ces techniques sont basées sur la détection du matériel moléculaire des microorganismes
- Hybridation fluorescente in situ (technique FISH) : utilisation de sondes oligonucléaires qui sont complémentaires et qui s’hybrident à des séquences d’ARNr ou ADN. L’hybridation est réalisée sur des cellules intactes, le marquage fait par des colorants fluorescents permet la détection par épifluorescence.
Pour en savoir plus sur la technique FISH : http://college-genetique.igh.cnrs.fr/Enseignement/genchrom/cytomol.html
- Hybridation fluorescente in situ (technique FISH) : utilisation de sondes oligonucléaires qui sont complémentaires et qui s’hybrident à des séquences d’ARNr ou ADN. L’hybridation est réalisée sur des cellules intactes, le marquage fait par des colorants fluorescents permet la détection par épifluorescence.
Pour en savoir plus sur la technique FISH : http://college-genetique.igh.cnrs.fr/Enseignement/genchrom/cytomol.html
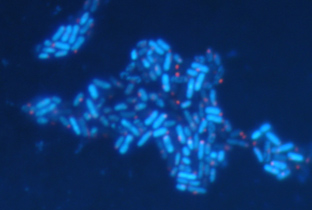
Figure 34: EPI fluorescence de bactéries Les bâtonnets bleus sont les bactéries qui lorsqu’elles sont vivantes ont un point rouge. (Source : CNRS photothèque / LETT Marie-Claire)
- La PCR (Polymérase Chain Reaction) est une technique permettant d’amplifier la présence d’ADN dans un échantillon pour en faciliter la détection. Cette amplification est faite en introduisant des séquences primaires complémentaires et spécifiques de la chaine d’ADN que l’on souhaite amplifier. En quelques heures on peut multiplier par un facteur 10 (6) l’ADN cible et ainsi mettre en évidence le microorganisme recherché.
Pour en savoir plus sur la PCR : http://www.bibliomer.com/documents/fiches/fiche_ensavoirplus_lien_PCR_vf.pdf
Click-Mediated Labeling of Bacterial Membranes through Metabolic Modification of the Lipopolysaccharide Inner Core : nouvelle technique développée pour détecter les bactéries vivantes.
La méthode mise au point permet de détecter les bactéries vivantes de type Gram négatif (ex : Escherichia coli, Salmonella typhimurium et Legionella pneumophila).Méthode :
- les bactéries sont mises en contact avec du KDO, un sucre dont les bactéries se servent pour synthétiser un polysaccharide spécifique de leur membrane cellulaire. Ce sucre a été au préalable modifié par l'introduction d'une fonction azoture (constituée de trois atomes d'azote).
- leurrées, les bactéries intègrent le sucre artificiel à leur membrane.
- grâce à une molécule fluorescente s'attachant exclusivement au groupe azoture, il devient alors possible de reconnaître et compter les bactéries Gram négatif vivantes, les seules à avoir assimilé le KDO modifié.Pour la suite, l'utilisation d'un sucre spécifique de chaque bactérie d'intérêt devrait permettre la détection d'un très large éventail de bactéries pathogènes vivantes.
Source : http://www2.cnrs.fr/presse/communique/2461.htm
Principale limite des méthodes moléculaires : il n’est pas possible de déterminer la viabilité des microorganismes. Hormis pour la technique ci-dessus
Pour en savoir plus sur la PCR : http://www.bibliomer.com/documents/fiches/fiche_ensavoirplus_lien_PCR_vf.pdf
Click-Mediated Labeling of Bacterial Membranes through Metabolic Modification of the Lipopolysaccharide Inner Core : nouvelle technique développée pour détecter les bactéries vivantes.
La méthode mise au point permet de détecter les bactéries vivantes de type Gram négatif (ex : Escherichia coli, Salmonella typhimurium et Legionella pneumophila).Méthode :
- les bactéries sont mises en contact avec du KDO, un sucre dont les bactéries se servent pour synthétiser un polysaccharide spécifique de leur membrane cellulaire. Ce sucre a été au préalable modifié par l'introduction d'une fonction azoture (constituée de trois atomes d'azote).
- leurrées, les bactéries intègrent le sucre artificiel à leur membrane.
- grâce à une molécule fluorescente s'attachant exclusivement au groupe azoture, il devient alors possible de reconnaître et compter les bactéries Gram négatif vivantes, les seules à avoir assimilé le KDO modifié.Pour la suite, l'utilisation d'un sucre spécifique de chaque bactérie d'intérêt devrait permettre la détection d'un très large éventail de bactéries pathogènes vivantes.
Source : http://www2.cnrs.fr/presse/communique/2461.htm
Principale limite des méthodes moléculaires : il n’est pas possible de déterminer la viabilité des microorganismes. Hormis pour la technique ci-dessus